宝塔怎么安装wordpress湖南seo服务
面临的问题
目前, 大数据计算引擎主要用 Java 或是基于 JVM 的编程语言实现的,例如 Apache Hadoop、 Apache Spark、 Apache Drill、 Apache Flink 等。 Java 语言的好处在于程序员不需要太关注底层内存资源的管理,但同样会面临一个问题, 就是如何在内存中存储大量的数据(包括缓存和高效处理)。 Flink 使用自主的内存管理,来避免这个问题。
JVM 内存管理的不足
1) Java 对象存储密度低。
Java 的对象在内存中存储包含 3 个主要部分:对象头、实例数据、对齐填充部分。例如, 一个只包含 boolean 属性的对象占 16byte:对象头占 8byte,
boolean 属性占 1byte, 为了对齐达到 8 的倍数额外占 7byte。而实际上只需要一个 bit(1/8
字节)就够了。
2) Full GC 会极大地影响性能。
尤其是为了处理更大数据而开了很大内存空间的 JVM来说, GC 会达到秒级甚至分钟级。
3) OOM 问题影响稳定性。
OutOfMemoryError 是分布式计算框架经常会遇到的问题,当JVM中所有对象大小超过分配给JVM的内存大小时,就会发生OutOfMemoryError错误,导致 JVM 崩溃,分布式框架的健壮性和性能都会受到影响。
4)缓存未命中问题。
CPU 进行计算的时候,是从 CPU 缓存中获取数据。 现代体系的 CPU 会有多级缓存,而加载的时候是以 Cache Line 为单位加载。如果能够将对象连续存储,这样就会大大降低 Cache Miss。使得 CPU 集中处理业务,而不是空转。(Java 对象在堆上存储的时候并不是连续的,所以从内存中读取 Java 对象时,缓存的邻近的内存区域的数据往往不是 CPU 下一步计算所需要的,这就是缓存未命中。 此时 CPU 需要空转等待从内存中重新读取数据。)Flink 并不是将大量对象存在堆内存上,而是将对象都序列化到一个预分配的内存块上,这个内存块叫做 MemorySegment,它代表了一段固定长度的内存(默认大小为 32KB),也是 Flink 中最小的内存分配单元,并且提供了非常高效的读写方法,很多运算可以直接操作二进制数据,不需要反序列化即可执行。每条记录都会以序列化的形式存储在一个或多个MemorySegment 中。 如果需要处理的数据多于可以保存在内存中的数据, Flink 的运算符会将部分数据溢出到磁盘。
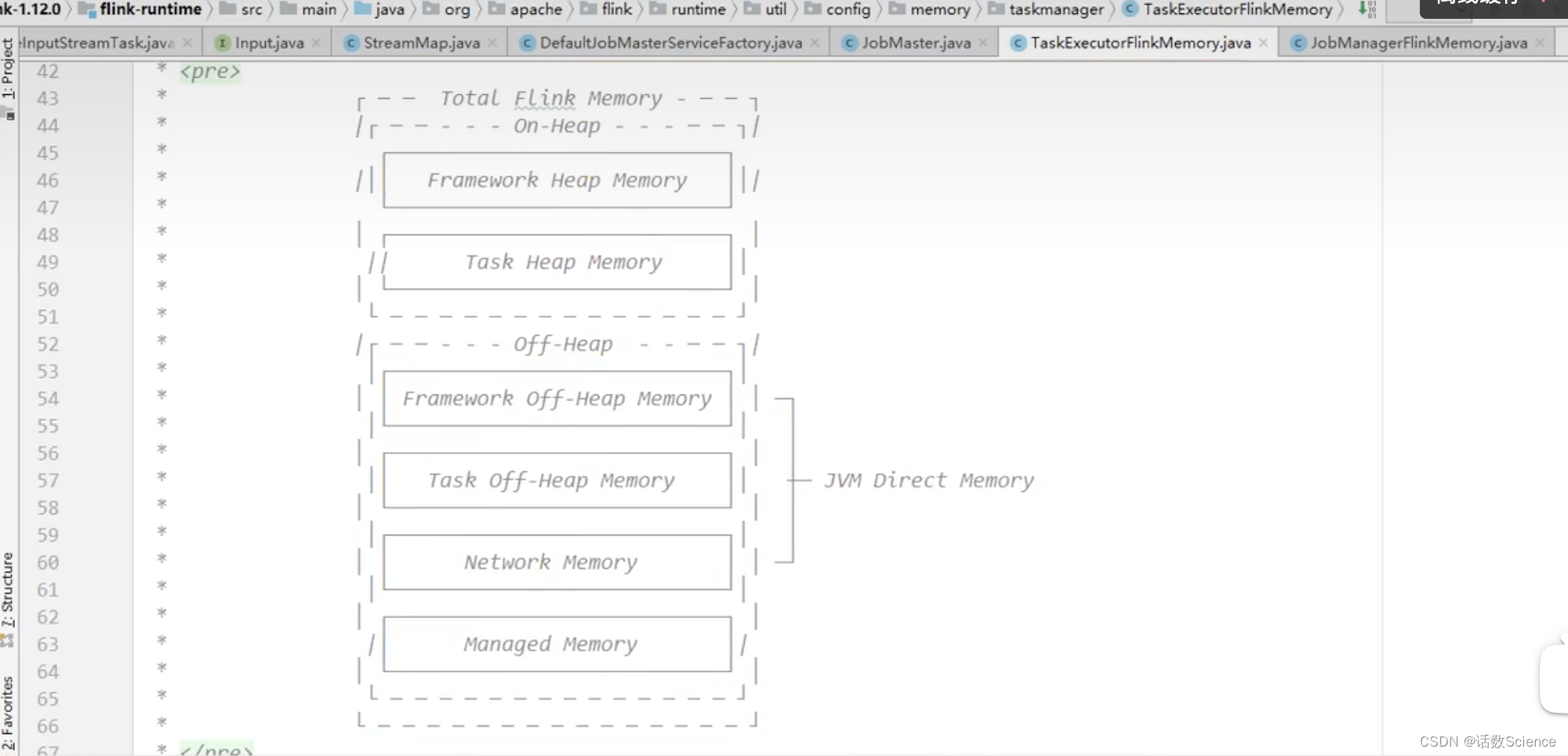
JobManager内存模型

TaskManager内存模型

内存结构
内存段
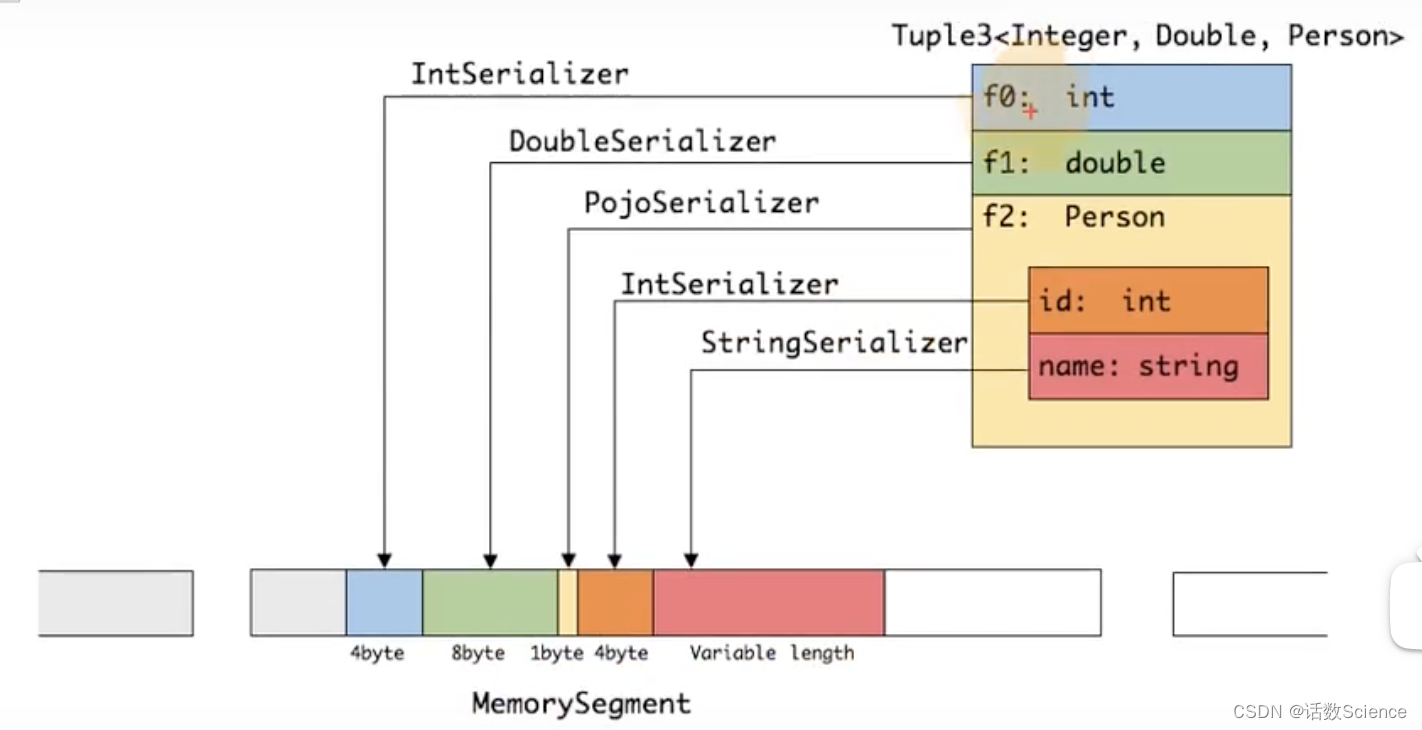
可以看出这种序列化方式存储密度是相当紧凑的。其中 int 占 4 字节, double 占 8 字
节, POJO 多个一个字节的 header, PojoSerializer 只负责将 header 序列化进去,并委托每个
字段对应的 serializer 对字段进行序列化。
内存页
内存页是 MemorySegment 之上的数据访问视图, 数据读取抽象为 DataInputView,
数据写入抽象为 DataOutputView。使用时就无需关心 MemorySegment 的细节,会自
动处理跨 MemorySegment 的读取和写入。
Buffer
Task 算子之间在网络层面上传输数据, 使用的是 Buffer, 申请和释放由 Flink
自行管理, 实现类为 NetworkBuffer。 1 个 NetworkBuffer 包装了 1 个
MemorySegment。 同时继承了 AbstractReferenceCountedByteBuf, 是 Netty 中的抽
象类。
网络缓存
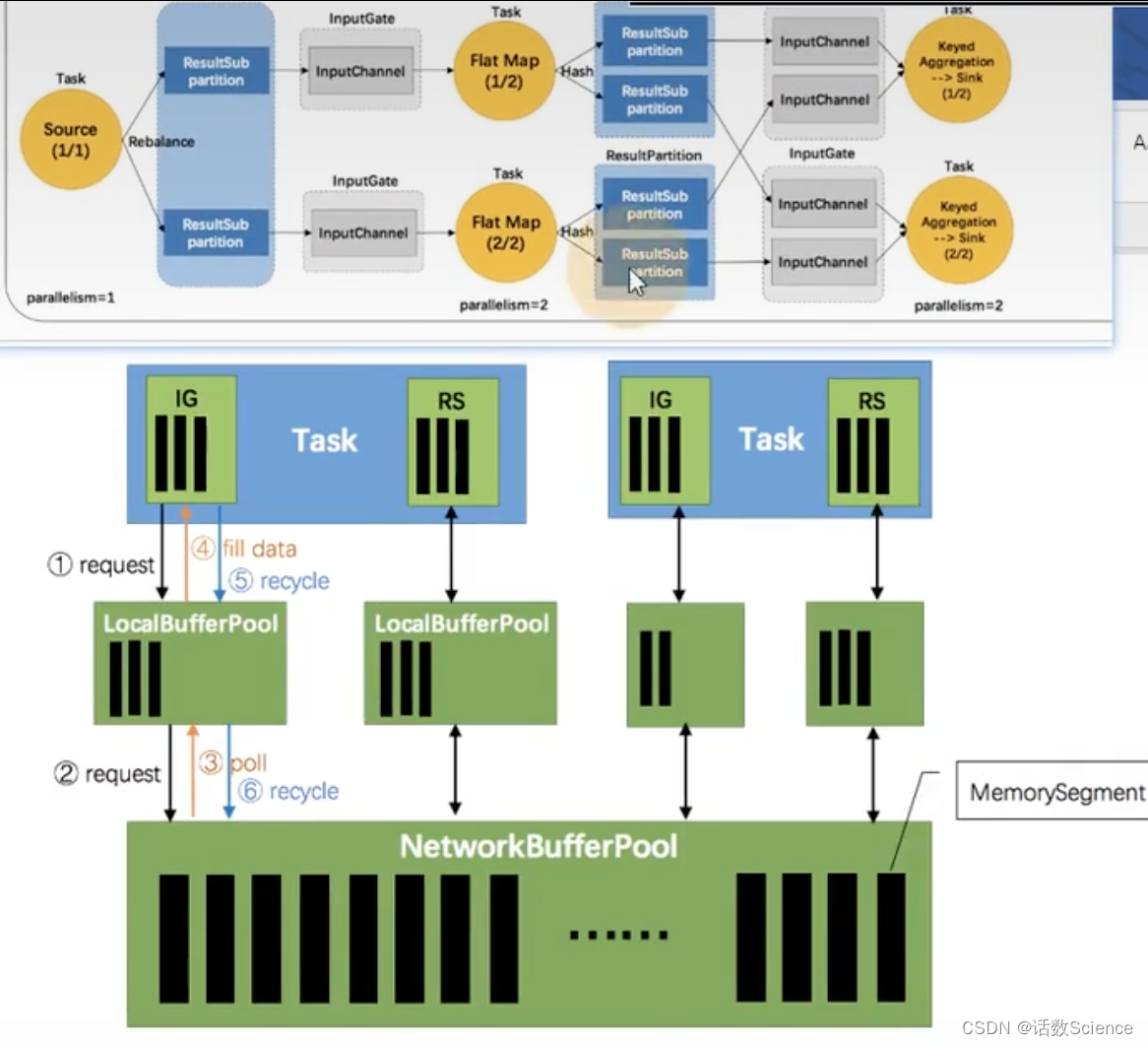
对照物理执行计划
IG:input gate
RS:结果分区